diseaseTag - Extraction d’entités nommées de maladies
Ce web service détecte des entités nommées de maladies sur des textes en anglais.
Les entités nommées sont toutes renvoyées dans un champ “diseases”.
Le modèle utilisé par ce web service, “bio-ner”, est disponible à ce lien huggingface.
Aucun fine-tuning n’a été opéré : nous mettons simplement le modèle à disposition via un web service.
Le code git pour fine-tuner le modèle est disponible à ce lien.
Le modèle possède une f-mesure de 0.8 ± 0.04.
Précaution : Le web service fonctionne uniquement sur du texte anglais.
- Article explicitant la méthodologie pour obtenir le modèle bio-ner : Alonso Casero, Álvaro (2021). Named entity recognition and normalization in biomedical literature: a practical case in SARS-CoV-2 literature. Thesis (Master thesis), E.T.S. de Ingenieros Informáticos (UPM)
- Le modèle bio-ner est obtenu après fine-tuning du modèle bioBERT : Data and text mining. BioBERT: a pre-trained biomedical language representation model for biomedical text mining
Utilisation dans TDM Factory
Ce web service se lance sur :
- un corpus en anglais Istex au format targz
- un document en anglais au format PDF texte (le format PDF image ne fonctionne pas)
Utilisation dans Lodex
Sélectionnez le web service dans le catalogue :
Enrichissement : le web service traite chaque document l’un après l’autre. Exemple pour l’extraction de termes à partir du résumé. On parlera de web service synchrone.
Saisir cette URL https://diseases-ner.services.istex.fr/v1/diseases/tagger et sélectionner la colonne dédiée au texte en anglais.
Sauvegarder et lancer le traitement
Pour récupérer la valeur souhaitée : GET et le nom du champ dont vous voulez extraire la donnée : diseases
| They have been used in the prevention and treatment of malaria and autoimmune diseases, including systemic lupus erythematosus and rheumatoid arthritis. | ==> | malaria, autoimmune diseases, systemic lupus erythematosus, rheumatoid arthritis |
| This sentence does not contain any diseases named entities | ==> |
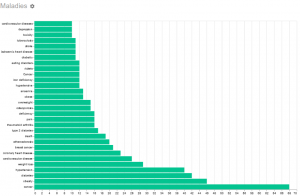
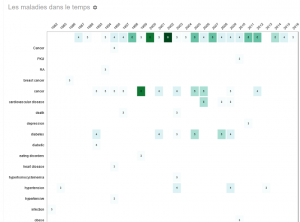
Vous avez un corpus consacré à une thématique qui peut être en lien avec la santé et vous souhaitez en connaître le contenu de manière plus précise ?
En lançant diseaseTag sur vos données depuis Lodex, logiciel libre de visualisation, vous obtiendrez les termes de maladies et des représentations graphiques liées.
Graphes créés à partir des données du corpus scientifique “Végane & Végétarien : Evolution des aspirations des personnes véganes et végétariennes au cours du temps”.
Profitez en pour naviguer dans l’ensemble du corpus
et découvrez d’autres corpus scientifiques